修回日期: 2020-06-12
接受日期: 2020-09-28
在线出版日期: 2020-11-28
炎症性肠病(inflammatory bowel disease, IBD)发病机制复杂, 可能与遗传因素、环境因素、肠黏膜屏障功能、免疫反应和肠道菌群有关, 且目前尚无可根治的药物, 亟需更多治疗药物的研究. 肠道菌群被认为是IBD发病机制的重要因素和IBD治疗药物研究的重要靶点, 其可以调节宿主的生命活动、调节免疫反应和抵御肠道菌群失调, 发挥保护宿主健康的重要作用. 本文对基于肠道菌群的IBD治疗药物研究策略进行了讨论, 人体相关动物模型和粪便移植的应用可有效阐明IBD治疗药物的作用机制, 可富集益生菌或抑制有害菌的药物具有巨大的治疗IBD的潜力, 结合多种分子技术探讨肠道菌群改变与药物治疗IBD的因果关系是未来IBD治疗药物研究的关注重点.
核心提要: 肠道菌群被认为是炎症性肠病(inflammatory bowel disease, IBD)发病机制的关键影响因素, 具有抵抗肠道菌群失衡和调节宿主基本生命活动和免疫反应的作用. 可富集益生菌或抑制有害菌的药物具有巨大的治疗IBD的潜力, 而利用人体菌群相关动物模型、粪便移植技术、多组学技术和微生物分子技术等可有效地阐明肠道菌群改变和药物治疗IBD的因果关系, 是未来IBD治疗药物研究的重点领域.
引文著录: 廖紫琼, 纪秋凤, 周本杰. 炎症性肠病的发病机制及基于肠道菌群的药物研究策略. 世界华人消化杂志 2020; 28(22): 1112-1120
Revised: June 12, 2020
Accepted: September 28, 2020
Published online: November 28, 2020
The pathogenesis of inflammatory bowel disease (IBD) is complex, which may be associated with genetic factors, environmental factors, intestinal mucosal barrier function, immune response, and gut microbiota. At present, there is no cure for IBD, and therefore there is an urgent need to develop therapeutic drugs. Gut microbiota is considered an important factor in the pathogenesis of IBD and is thus an important target for IBD drug research, with the function of regulating the vital activities of host, modulating the immune response, and protecting against intestinal flora disorders. This paper briefly discusses the strategies for IBD drug research by targeting gut microbiota. Fecal transplantation and human microbiota-associated model are effective ways to elucidate the mechanism of IBD therapeutic drugs. Drugs that can enrich probiotics or inhibit harmful bacteria have great potential for the treatment of IBD. Exploring the causal relationship between the changes in gut microbiota and IBD therapeutic drugs through multiple molecular techniques is the focus of IBD drug research in the future.
- Citation: Liao ZQ, Ji QF, Zhou BJ. Strategies for inflammatory bowel disease drug research by targeting gut microbiota. Shijie Huaren Xiaohua Zazhi 2020; 28(22): 1112-1120
- URL: https://www.wjgnet.com/1009-3079/full/v28/i22/1112.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v28.i22.1112
炎症性肠病(inflammatory bowel disease, IBD)是慢性的反复发作的肠道炎症性疾病, 患者常常在小肠和大肠黏膜处有持续性的溃疡, 主要分为两种类型, 溃疡性结肠炎(ulcerative colitis, UC)和克罗恩病(Crohn's disease, CD). 21世纪以前, IBD在西方国家的发病率比较高, 但随着工业化和城镇化发展, 其在亚太地区的发病率也逐渐上升. IBD在香港、台湾、澳门和中国大陆等地区的发病率均有上升的趋势, 在我国逐渐成为一种常见病[1-3]. 然而, 目前尚无可根治IBD的药物, IBD治疗药物的研究仍然需要广大科研人员的不断努力.
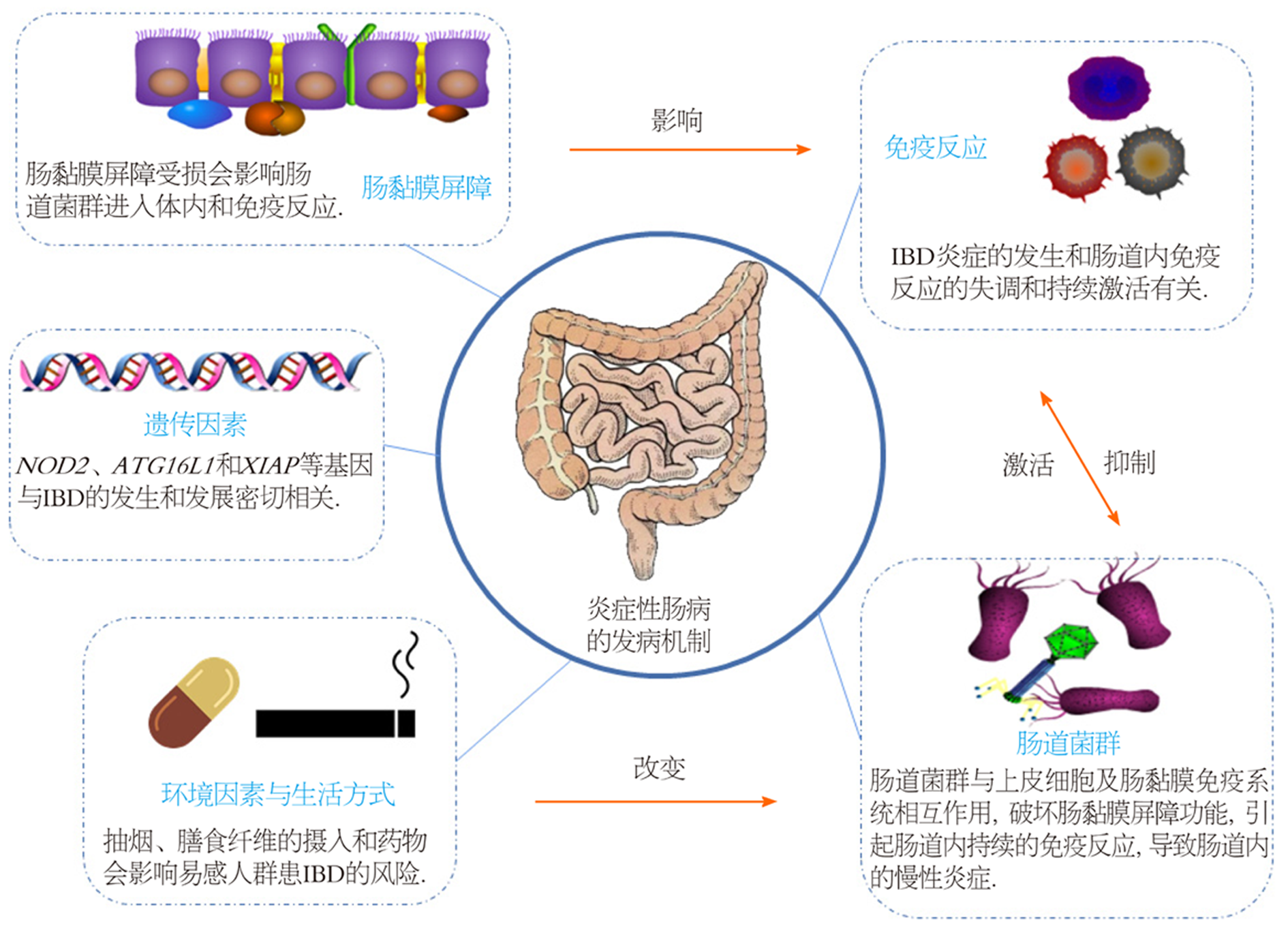
对IBD发病机制的理解是确定IBD治疗药物研究策略的关键. IBD的发病机制极其复杂, 可能与遗传易感性、环境因素、肠道菌群、肠黏膜功能、免疫、氧化应激和炎症等因素有关, 接下来我们将对IBD的发病机制和基于肠道菌群的IBD治疗药物研究策略进行详细阐述.
目前大量的全基因组关联研究表明, 像IBD这样复杂的遗传相关的疾病是由多种基因多样性引起的, 具有多基因性. 目前已经发现超过200个与IBD相关的基因位点, 且其中70%的基因位点也存在于其他复杂的自身免疫性疾病和免疫缺陷性疾病[4]. 增加CD易感性的遗传变异主要和先天免疫、自噬和吞噬有关, 而与UC易感性相关的遗传变异主要与肠屏障功能有关[5,6]. 已知16号染色体上的核苷酸结合寡聚化结构域蛋白2基因(nucleotide-binding oligomerization-2, NOD2, 又被称为CARD15和IBD1)是第一个被发现的与IBD密切相关的基因[5], NOD2基因的激活被证明会影响组织相容性复合体的交叉表达、自噬的诱导和对细胞内细菌感染的抵抗力[7-9]. 在自噬途径中发挥关键作用的自噬相关基因16L1(autophagy related 16 like 1, ATG16L1)和免疫相关鸟苷三磷酸酶基因(immunity-related GTPase family M protein, IRGM)的多态性与IBD也有密切联系[8,9], 在自噬过程中要实现有效的细胞内消化和细菌清除, NOD2和ATG16L1功能的完整性都是必需的[10]. 也有研究发现X染色体的连锁的凋亡抑制蛋白(X chromosome linked inhibitor of apoptosis protein, XIAP)的基因突变与难以治疗的早发性IBD有关[11]. 此外, 炎症因子23/辅助性T 细胞17(interleukin 23/T helper cell 17, IL-23/Th17)信号通路中的许多基因, 如白介素23受体基因(interleukin 23, IL23R)、白介素12B基因(interleukin 12B, IL12B), 酪氨酸激酶(Janus activated kinase, JAK3)和信号转换器和转录因子的激活剂3(Signal transducer and activator of transcription 3, STAT3)和IBD的发展有关[5,12].
虽然IBD的发病机制和遗传因素有关, 但目前仅有25%的IBD遗传可能性被遗传研究阐明[13], 且本流行于发达国家的IBD在工业化后在发展中国家也出现了流行的趋势, 由此可见, 环境因素和生活方式可能在促进IBD遗传易感人群患IBD中发挥着重要作用.
吸烟被认为是IBD的独立高危因素[14], 但也有研究表明吸烟与IBD没有联系, 产生这些矛盾的结果的原因仍需要我们进一步研究[15]. 据报道, 富含饱和脂肪酸的饮食和加工肉类的过多摄入会增加患IBD的风险[16,17], 相反, 高纤维饮食人群患CD的风险会降低40%, 这种现象可能与膳食纤维可被肠道菌群代谢成具有抗炎作用的短链脂肪酸(short-chain fatty acids, SCFAs)有关[18]. 此外, 一些药物的使用, 如非甾体类抗炎药、避孕药、他汀类药物和抗生素等, 也会增加易感人群患IBD的风险[19-22]. 这些环境因素和生活方式对IBD的影响被认为可能和肠道菌群组成的破坏有关.
肠黏膜屏障由机械屏障、化学屏障、免疫屏障与生物屏障共同构成. 上世纪70年代, 首先有科学家提出, CD的发生可能与肠黏膜屏障受损和功能发生异常有关, 之后的研究也逐渐证实肠黏膜屏障异常和受损可增加肠道菌群等抗原通过肠黏膜的机会, 引起肠道免疫的过度反应, 导致持续的慢性炎症[23,24]. 现有研究报道, 与正常人相比, IBD患者肠上皮细胞通透性增加和肠黏膜屏障功能受损, 且在CD患者复发前可观察到肠道通透性增加的现象[25-27]. 此外, 有研究报道CD患者和IBD模型动物的肠道紧密连接蛋白(如Claudin-1蛋白、Occludin蛋白和ZO-1蛋白)的含量会下调, 且与肠上皮再生相关的转录因子, 如同源框蛋白质NKX2-3和肝细胞核因子4, 也被报道与IBD有联系[28,29]. 肠黏膜屏障功能受损可能会导致肠道内促进肠道菌群和肠上皮细胞分离、杀害渗透性肠道菌群和抑制过度免疫反应的功能出现紊乱, 肠道发生持续的免疫反应, 从而致肠道内发生持续性的慢性炎症. 因此, 肠黏膜屏障功能的受损和肠道菌群的分离杀害在IBD的发生和发展中发挥着关键作用[30].
IBD与免疫反应的紊乱有关, 且不同类型和程度的IBD的免疫失调存在差异, 如CD患者肠道黏膜上的T细胞主要是辅助性T细胞1(T helper 1 cell, Th1细胞), 而UC患者肠道黏膜上的T细胞主要是辅助性T细胞2(T helper 2 cell, Th2细胞), 可以分泌转化生长因子-β(transforming growth factor-β, TGF-β)和IL-5[31,32]. 可分泌大量IL-17A、IL-17F、IL-21和IL-22等细胞因子的Th17细胞也被认为可影响IBD的发生和发展[33]. Tregs细胞可以维持肠道内稳态, 因此也有研究者提出把Tregs细胞作为控制IBD过度炎症的替代策略[34,35]. 此外, IBD患者体内还存在细胞因子的失衡, 导致体内免疫反应的持续激活, 目前正在开发的IBD免疫疗法也主要是对不同细胞因子相关通路的下游通路的阻断和抑制, 从而减轻IBD的炎症, 如JAKs和信号转换器和STATs[36]. 因此, IBD炎症的发生和肠道内免疫反应的失调和持续激活有关. 免疫反应失调导致肠道炎症的过程可简单理解为: 肠道菌群等抗原入侵并通过肠黏膜屏障, 遗传易感人群体内树突状细胞等抗原递呈细胞发起一系列的促炎和抗炎信号, 从而激活不同区域的免疫细胞亚群并促使循环淋巴细胞向肠道发生炎症的位点迁移, 引起肠道的炎症, 且抗炎和促炎因子进一步失调并导致肠道内持续的炎症[37-40]. 由此可见, 免疫反应是IBD重要的发病因素和药物研究的重要靶点, 肠道菌群在引起持续性免疫反应中发挥着不可忽视的作用.
肠道菌群被认为是连接外界环境和肠道黏膜的纽带, 其和宿主防御反应之间的动态平衡对IBD的发生和发展产生影响[41]. 目前研究表明, IBD患者及该疾病模型中均出现肠道菌群失调的现象, 其变化主要表现出以下几个共性特征[42-45]: (1)肠道菌群的生物多样性降低: 16S rRNA和宏基因组测序的结果均显示IBD患者体内肠道菌群的种类显著少于正常健康者; (2)有益菌的相对丰度减少: IBD患者体内两个主要的菌群, 拟杆菌门(Bacteroidetes)和厚壁菌门(Firmicutes)的相对丰度显著降低; (3)潜在致病菌相对丰度升高: 在IBD患者体内可以发现变形菌门(Proteobacteria)和放线菌门(Actinobacteria)的相对丰度明显增加. 其中部分变形菌门的菌群已被报道与IBD密切相关, 如肠杆菌科(Enterobacteriaceae)的大肠杆菌(Escherichia coli)、产碱菌科(Alcaligenaceae)的萨特菌属(Sutterella)和脱硫弧菌科(Desulfovibrionaceae)的脱硫弧菌属(Desulfovibrio)等.
通过上文可知, 环境因素和生活方式可通过改变肠道菌群的组成影响IBD的发生和发展, 且肠道菌群与宿主的上皮细胞及肠黏膜免疫系统相互作用, 破坏肠黏膜屏障的功能, 引起遗传易感人群肠道内持续的免疫反应, 从而导致肠道内的慢性炎症[6,46], 图1. 由此可知, 肠道菌群是IBD的发病关键因素, 也是IBD治疗药物研究的重要靶点.
肠道菌群是IBD治疗药物研究的重要靶点, 正确认识肠道菌群在宿主体内发挥的关键作用有助于对IBD进行有选择性和有针对性的干预, 为IBD治疗药物的研究提供理论依据.
进入结肠未被宿主利用的碳水化合物、黏蛋白或者宿主的分泌物都可以被肠道菌群利用, 产生SCFAs、其他有机酸和二氧化碳等产物, 影响机体的能量代谢、肠道黏膜的正常功能和新陈代谢等生命活动. 首先, 基因测序结果显示, 肠道菌群中含有许多与蔗糖、淀粉、多糖、阿拉伯糖、甘露糖、木糖、异种化合物代谢相关的基因. 也有研究发现IBD患者体内肠道菌群对碳水化合物的代谢和利用能力下降, 而小檗碱干预可以改善肠道菌群的该功能, 并调节宿主能量代谢[47,48]. 同时, 厚壁菌门(Firmicutes)等肠道菌群可对碳水化合物进行代谢产生SCFAs, 调节肠道的酸碱平衡, 参与肠道内多种营养物质的吸收、激素的产生与调控和宿主的能量代谢等生命活动[49-51]. 再者, 肠道菌群的氨基酸代谢也可参与肠道稳态的维持, 色氨酸可代谢成芳香羟受体(Aryl hydrocarbon receptor, AhR)的配体, 发挥促进肠上皮细胞更新和保护肠黏膜的完整性的作用, 而IBD患者体内肠道菌群对色氨酸的代谢能力明显下降, 表现出明显的代谢失调[16,52-55]. 此外, 肠道菌群对胆汁酸的代谢产物可以反过来影响肠道菌群和机体相关基因的表达, 从而影响机体的生命活动[56]. 总而言之, 在宿主体内肠道菌群可以通过特有的菌群结构、活动及代谢产物等影响着机体的生理活动以及新陈代谢, 从而维持体内环境的稳态, 而IBD患者体内出现肠道菌群结构和代谢的失调. 因此, 肠道菌群对宿主生命活动的调节也是IBD治疗药物潜在的作用机制.
肠道菌群与宿主的免疫反应也密切相关. 肠道内成千上万的肠道菌群可以产生大量的有害物质、配体和抗原, 如脂多糖等, 这些物质可能影响肠黏膜的功能, 激活致病性先天免疫反应和适应性免疫反应, 从而引起一系列的持续性的免疫反应[57-59]. 同时, 肠道菌群可能参与宿主免疫系统的发育, 已知无菌小鼠全身免疫系统发育不全, 固有层、肠系膜淋巴结和脾脏的细胞结构减少, 黏膜免疫功能下降, 然而, 在无菌小鼠上重建肠道菌群可以一定程度上恢复无菌小鼠的免疫能力, 由此可见, 免疫系统的成熟与肠道菌群的完整密不可分[60-62]. 此外, 肠道菌群的代谢产物SCFAs对免疫细胞Tergs细胞具有调节作用, 且Th17细胞的激活也受到肠道菌群的影响[48,63]. 有研究发现, IBD患者体内乳杆菌属(Lactobacillus)可以代谢色氨酸产生吲哚乙酸等有效活性物质并调节IL-22的分泌, 从而减轻肠道的炎症[54]. 色氨酸代谢物对宿主免疫的调节可能与AhR、核因子κB和磷酸肌醇3-激酶等免疫信号通路的激活有关[60,64]. 总而言之, 肠道菌群可引起肠道的持续性免疫反应, 对免疫系统的发育具有重要调节作用, 基于肠道菌群的IBD治疗药物的研究应关注肠道菌群对免疫反应的调节作用.
稳定的肠道菌群有助于宿主防御病原体, 肠道内共生菌可竞争性地阻止病原菌的入侵, 这种防御机制称为"定植抗性"[65]. 研究表明, 无菌环境下的动物更易受到肠道病原体的感染, 这可能与免疫系统的异常或"定植抗性"的缺失有关. 此外, 肠道菌群可以通过直接或间接的作用机制增强对肠道病原体的抵抗作用: (1)通过争夺营养物质, 如多形拟杆菌(Bacteroides thetaiotaomicron)可通过消耗柠檬酸杆菌(Citrobacter rodentium)所需的碳水化合物从而实现抑制作用; (2)通过分泌抑制物质直接抑制肠道病原体[66], 如苏云金杆菌(Bacillus thuringiensis)可分泌一种针对杆菌和梭状芽胞杆菌的细菌素竞争性地抑制艰难梭杆菌(Clostridium difficile)的定植[67]; (3)通过激活免疫反应间接抵御病原体, 肠道菌群的共生菌群及其产物, 如脂多糖和鞭毛蛋白等, 可激活免疫反应并通过免疫反应间接抵御和清除病原体[68,69]. 肠道的共生菌通过"定植抗性"发挥抵御病原体和保护宿主健康的重要作用, 因此在IBD治疗药物研究过程中应关注肠道菌群共生菌的稳定性和肠道菌群稳态的重构.
在IBD治疗药物的研究过程中, 常用动物模型探讨药物对IBD的治疗效果和作用机制. 用于IBD研究的动物模型有许多, 其各有优缺点, 主要分为3大类[48,70-73]: (1)自发性肠炎模型, 自然界动物自然发生或近亲繁殖形成的自发性的肠炎, 如SAMPl/Yit小鼠和C3H/HejBir小鼠可产生和人类IBD相似的肠道炎症, 但该类模型比较稀少和昂贵, 且实验过程中难以控制炎症的发展进程; (2)诱导产生的肠炎模型, 主要分为化学试剂诱导的肠炎[如2,4,6-三硝基苯磺酸(TNBS)、葡聚糖硫酸钠(DSS)、醋酸]、病原菌诱导的肠炎和免疫诱导的肠炎, 该类模型相对比较稳定, 可用于IBD药物疗效和发病机制的研究[48,73,74]; (3)基因改造动物模型, 指通过转基因或条件性基因剔除等技术建立的肠炎模型, 如白介素10(Interleukin 10, IL-10)基因敲除的小鼠可产生和IBD相似的症状, 该类模型可用于研究特定基因的功能和调控机制, 但模型的建立耗时长, 成本高[75].
IBD治疗药物的研究常用到以上这些动物模型, 但这些模型在基于肠道菌群的IBD治疗药物研究上均存在一定的局限性. 由于以上模型的建立方法机制不一, 且人类和动物的肠道菌群也不完全一致, 所以以上动物模型的肠道菌群和IBD患者的肠道菌群会存在一定的差异, 无法充分研究IBD患者在治疗过程中肠道菌群的变化及发挥的作用. 因此, 在进行肠道菌群相关研究的时候, 可将粪便中的肠道菌群从有或没有疾病或病症的个体移植到无菌的啮齿动物中, 建立相应的模型, 然后对受体动物的病理表型进行比较, 并进一步研究药物疗效和作用机制. 这种模型叫做人体菌群相关动物模型, 可以更好地阐述肠道菌群和疾病、药物疗效的因果关系, 是在确立肠道菌群改变与人类自身疾病的因果关系的研究中的金标准和基石. 有研究者利用人体菌群相关小鼠模型将健康人和IBD患者的肠道菌群定植于无菌小鼠上进行研究, 发现IBD患者肠道菌群的定植导致Th17细胞和Th2细胞数量的增加和RORγt + Treg细胞数量的减少[76]. Gobert等[77]人利用人体菌群相关动物模型发现了特发性肠易激综合征患者的肠道菌群可能通过嗜黏蛋白艾克曼菌(Akkermansia muciniphila)对DSS诱导的肠炎发挥抗炎作用. 由此可见, 人体菌群相关动物模型可以有效应用于IBD与肠道菌群相关的潜在发病机制和相关治疗药物的研究.
肠道菌群的稳定对维持宿主的健康十分重要. 进行粪便移植可以重塑肠道内稳定的肠道菌群. 现在的研究也表明, 粪便移植在与肠道菌群具有明确关系的疾病如艰难梭状杆菌感染的治疗有良好的疗效. 此外, 粪便移植也可应用于基于肠道菌群的IBD治疗药物的研究, 将药物治疗后的粪便移植至IBD模型动物中, 可验证药物对IBD的疗效是否通过肠道菌群的重塑发挥作用, 有效地探讨肠道菌群在疾病治疗过程中扮演的角色. 例如, 有研究者将小檗碱治疗UC后的粪便移植至UC小鼠中, 证明了小檗碱可通过肠道菌群调节Th17细胞并缓解UC的症状[78]. 目前, 粪便移植在IBD治疗药物的研究中得到了广泛应用. 然而, 由于"定植抗性"的存在, 粪便移植过程中肠道菌群是否能在受体中成功定植也是进行IBD治疗药物研究过程中需要关注的一个问题, 而将粪便移植至无菌小鼠或利用抗生素建立的伪无菌小鼠上可减少肠道菌群"定植抗性"的影响[79-81]. 在粪便移植之前使用抗生素可能会增加受体耐药菌定植的几率, 而在粪便移植时将剂量提高两倍有利于耐药菌在受体的去定植. 此外, 大部分肠道菌群是厌氧菌, 在粪便移植中, 粪便置于空气中操作和反复冻融也可能降低肠道菌群的定植率, 无菌操作可以提高供体肠道菌群的定植率[82]. 虽然粪便移植发生重大不良反应的几率较低, 但还是会存在一定的风险, 其可能会导致供体致病因子的感染, 如耐药菌和供体接种疫苗后可能还停留在粪便中的脊髓灰质病毒等[83]. 因此, 在进行粪便移植时需要对供体进行严格的微生物筛查并建立标准化的流程, 特别是应用于人体的临床试验时.
随着高通量测序技术的发展, IBD患者体内肠道菌群的失衡已经被广泛报道[44,84,85]. 在IBD治疗药物的研究中应关注药物与和IBD密切相关的肠道菌群的关系, 鉴定出发挥关键作用的肠道菌属. 此外, 靶向这些关键的异常肠道菌属, 如减少致病性强的菌属和提高有益菌属的浓度和代谢活性, 可以提高药物治疗IBD的潜力. 目前抗生素、益生菌和能刺激益生菌生长的益生元等应用于IBD的治疗已得到广泛的报道[47,86,87]. 可抑制有害菌的利福平、克林霉素和小檗碱等广谱抗生素对IBD模型动物具有良好的保护作用; 双歧杆菌属(Bifidobacterium)、乳杆菌属(Lactobacillus)和柔嫩梭菌属(Faecalibacterium)等益生菌已被证实可以减轻IBD的炎症; 一些复合多糖、低聚果糖和菊粉等益生元则可保护益生菌的生长和代谢, 起到与益生菌相似的疗效[78,88-90]. 益生菌的富集和有害菌的抑制可能通过调节炎症因子的分泌和代谢影响肠道的免疫反应和肠道黏膜屏障的功能等发挥改善IBD的作用, 如有研究发现柔嫩梭菌属(Faecalibacterium)与CARD9基因(胱天蛋白酶募集域蛋白9, Caspase recruitment domain 9)共同影响色氨酸的代谢, 并调节免疫系统和免疫因子IL-22的表达, 从而影响IBD的发生和发展[54]. 这些抗生素、益生菌和益生元组成的复合物可有效地抑制病原菌且促进益生菌的生长, 是良好的治疗IBD的潜在药物.
基于肠道菌群的IBD治疗药物研究中, 许多科研人员利用基因组学、代谢组学和蛋白组学等技术研究药物对IBD的治疗效果、肠道菌群结构和功能的影响, 探讨关键分子(肠道菌属、代谢分子和蛋白等)与药物对IBD疗效的相关性[74,78]. 然而, 目前较多研究仅立足于药物对IBD疗效与肠道菌群变化的相关性研究, 药物疗效与肠道菌群、代谢物和蛋白的变化之间的联系究竟是因果关系还是伴随关系仍需要更多的研究, 比如上文提到的具有较大潜力的益生菌和益生元治疗IBD的具体作用机制的研究目前仍然比较欠缺, 且许多研究仍然停留在动物水平阶段, 仍需要更多深入的研究. 对肠道菌群的变化与药物疗效的因果关系进行探究可深入揭示疾病的发病机制和药物的作用机制. 例如, 有研究者结合多种技术揭示了CARD9基因的缺乏会导致肠道内乳杆菌属(Lactobacillus)等菌属无法代谢色氨酸产生AhR配体, 从而导致IBD的发生的发展, 探讨了肠道菌群的变化、宿主和基因多样化的直接联系, 也为IBD治疗药物的研究提供了新靶点[57]. 因此, 在未来IBD治疗药物的研究中, 我们需要关注和重视IBD治疗和肠道菌群变化的因果关系, 利用复杂的分子微生物学技术, 将肠道菌群的变化与宿主、基因多态性和基因表达等相结合, 探究药物治疗IBD过程中肠道菌群与宿主的相互作用, 明确肠道菌群发挥的关键作用, 深入探讨药物的治疗机制, 也为IBD的治疗提供更多可靠的治疗靶点.
IBD发病机制复杂, 目前尚无可根治的药物, 亟需更多药物的研究. 环境因素可影响肠道菌群和肠黏膜屏障的功能, 而肠道菌群的改变和致病病原菌的入侵可能诱发遗传易感人群肠道发生持续性免疫反应, 导致肠道内的慢性炎症. 此外, 肠道菌群具有抵制肠道菌群失调、调节宿主基本生命活动和免疫反应的作用, 是IBD治疗药物研究的重要靶点. 人体菌群相关动物模型和粪便移植的应用可更有效地阐明肠道菌群变化和药物治疗IBD的因果关系, 可富集益生菌或抑制有害菌的药物具有巨大的治疗IBD的潜力. 最后, 结合基因组学、代谢组学、蛋白质组学和分子微生物学等技术, 将药物治疗IBD过程中肠道菌群的变化与宿主、基因多态性和基因表达等相结合, 可探究肠道菌群与药物治疗IBD的因果关系和作用机制, 也是未来IBD治疗药物研究需重点关注的领域.
学科分类: 胃肠病学和肝病学
手稿来源地: 广东省
同行评议报告学术质量分类
A级 (优秀): 0
B级 (非常好): 0
C级 (良好): C, C
D级 (一般): 0
E级 (差): 0
科学编辑: 马亚娟 制作编辑: 张砚梁
| 1. | Ananthakrishnan AN. Epidemiology and risk factors for IBD. Nat Rev Gastroenterol Hepatol. 2015;12:205-217. [PubMed] [DOI] |
| 2. | Kuo CJ, Yu KH, See LC, Chiu CT, Su MY, Hsu CM, Kuo CF, Chiou MJ, Liu JR, Wang HW. The Trend of Inflammatory Bowel Diseases in Taiwan: A Population-Based Study. Dig Dis Sci. 2015;60:2454-2462. [PubMed] [DOI] |
| 3. | Yang H, Li Y, Wu W, Sun Q, Zhang Y, Zhao W, Lv H, Xia Q, Hu P, Li H, Qian J. The incidence of inflammatory bowel disease in Northern China: a prospective population-based study. PLoS One. 2014;9:e101296. [PubMed] [DOI] |
| 4. | Jostins L, Ripke S, Weersma RK, Duerr RH, McGovern DP, Hui KY, Lee JC, Schumm LP, Sharma Y, Anderson CA, Essers J, Mitrovic M, Ning K, Cleynen I, Theatre E, Spain SL, Raychaudhuri S, Goyette P, Wei Z, Abraham C, Achkar JP, Ahmad T, Amininejad L, Ananthakrishnan AN, Andersen V, Andrews JM, Baidoo L, Balschun T, Bampton PA, Bitton A, Boucher G, Brand S, Büning C, Cohain A, Cichon S, D'Amato M, De Jong D, Devaney KL, Dubinsky M, Edwards C, Ellinghaus D, Ferguson LR, Franchimont D, Fransen K, Gearry R, Georges M, Gieger C, Glas J, Haritunians T, Hart A, Hawkey C, Hedl M, Hu X, Karlsen TH, Kupcinskas L, Kugathasan S, Latiano A, Laukens D, Lawrance IC, Lees CW, Louis E, Mahy G, Mansfield J, Morgan AR, Mowat C, Newman W, Palmieri O, Ponsioen CY, Potocnik U, Prescott NJ, Regueiro M, Rotter JI, Russell RK, Sanderson JD, Sans M, Satsangi J, Schreiber S, Simms LA, Sventoraityte J, Targan SR, Taylor KD, Tremelling M, Verspaget HW, De Vos M, Wijmenga C, Wilson DC, Winkelmann J, Xavier RJ, Zeissig S, Zhang B, Zhang CK, Zhao H; International IBD Genetics Consortium (IIBDGC), Silverberg MS, Annese V, Hakonarson H, Brant SR, Radford-Smith G, Mathew CG, Rioux JD, Schadt EE, Daly MJ, Franke A, Parkes M, Vermeire S, Barrett JC, Cho JH. Host-microbe interactions have shaped the genetic architecture of inflammatory bowel disease. Nature. 2012;491:119-124. [PubMed] [DOI] |
| 5. | Barrett JC, Hansoul S, Nicolae DL, Cho JH, Duerr RH, Rioux JD, Brant SR, Silverberg MS, Taylor KD, Barmada MM, Bitton A, Dassopoulos T, Datta LW, Green T, Griffiths AM, Kistner EO, Murtha MT, Regueiro MD, Rotter JI, Schumm LP, Steinhart AH, Targan SR, Xavier RJ; NIDDK IBD Genetics Consortium, Libioulle C, Sandor C, Lathrop M, Belaiche J, Dewit O, Gut I, Heath S, Laukens D, Mni M, Rutgeerts P, Van Gossum A, Zelenika D, Franchimont D, Hugot JP, de Vos M, Vermeire S, Louis E; Belgian-French IBD Consortium; Wellcome Trust Case Control Consortium, Cardon LR, Anderson CA, Drummond H, Nimmo E, Ahmad T, Prescott NJ, Onnie CM, Fisher SA, Marchini J, Ghori J, Bumpstead S, Gwilliam R, Tremelling M, Deloukas P, Mansfield J, Jewell D, Satsangi J, Mathew CG, Parkes M, Georges M, Daly MJ. Genome-wide association defines more than 30 distinct susceptibility loci for Crohn's disease. Nat Genet. 2008;40:955-962. [PubMed] [DOI] |
| 6. | Xavier RJ, Podolsky DK. Unravelling the pathogenesis of inflammatory bowel disease. Nature. 2007;448:427-434. [PubMed] [DOI] |
| 7. | Strober W, Watanabe T. NOD2, an intracellular innate immune sensor involved in host defense and Crohn's disease. Mucosal Immunol. 2011;4:484-495. [PubMed] [DOI] |
| 8. | Hampe J, Franke A, Rosenstiel P, Till A, Teuber M, Huse K, Albrecht M, Mayr G, De La Vega FM, Briggs J, Günther S, Prescott NJ, Onnie CM, Häsler R, Sipos B, Fölsch UR, Lengauer T, Platzer M, Mathew CG, Krawczak M, Schreiber S. A genome-wide association scan of nonsynonymous SNPs identifies a susceptibility variant for Crohn disease in ATG16L1. Nat Genet. 2007;39:207-211. [PubMed] [DOI] |
| 9. | Rioux JD, Xavier RJ, Taylor KD, Silverberg MS, Goyette P, Huett A, Green T, Kuballa P, Barmada MM, Datta LW, Shugart YY, Griffiths AM, Targan SR, Ippoliti AF, Bernard EJ, Mei L, Nicolae DL, Regueiro M, Schumm LP, Steinhart AH, Rotter JI, Duerr RH, Cho JH, Daly MJ, Brant SR. Genome-wide association study identifies new susceptibility loci for Crohn disease and implicates autophagy in disease pathogenesis. Nat Genet. 2007;39:596-604. [PubMed] [DOI] |
| 10. | Homer CR, Richmond AL, Rebert NA, Achkar JP, McDonald C. ATG16L1 and NOD2 interact in an autophagy-dependent antibacterial pathway implicated in Crohn's disease pathogenesis. Gastroenterology. 2010;139:1630-1641, 1641.e1-1641.e2. [PubMed] [DOI] |
| 11. | Worthey EA, Mayer AN, Syverson GD, Helbling D, Bonacci BB, Decker B, Serpe JM, Dasu T, Tschannen MR, Veith RL, Basehore MJ, Broeckel U, Tomita-Mitchell A, Arca MJ, Casper JT, Margolis DA, Bick DP, Hessner MJ, Routes JM, Verbsky JW, Jacob HJ, Dimmock DP. Making a definitive diagnosis: successful clinical application of whole exome sequencing in a child with intractable inflammatory bowel disease. Genet Med. 2011;13:255-262. [PubMed] [DOI] |
| 12. | Franke A, Balschun T, Karlsen TH, Sventoraityte J, Nikolaus S, Mayr G, Domingues FS, Albrecht M, Nothnagel M, Ellinghaus D, Sina C, Onnie CM, Weersma RK, Stokkers PC, Wijmenga C, Gazouli M, Strachan D, McArdle WL, Vermeire S, Rutgeerts P, Rosenstiel P, Krawczak M, Vatn MH; IBSEN study group, Mathew CG, Schreiber S. Sequence variants in IL10, ARPC2 and multiple other loci contribute to ulcerative colitis susceptibility. Nat Genet. 2008;40:1319-1323. [PubMed] [DOI] |
| 13. | McGovern DP, Kugathasan S, Cho JH. Genetics of Inflammatory Bowel Diseases. Gastroenterology. 2015;149:1163-1176.e2. [PubMed] [DOI] |
| 14. | Agus A, Planchais J, Sokol H. Gut Microbiota Regulation of Tryptophan Metabolism in Health and Disease. Cell Host Microbe. 2018;23:716-724. [PubMed] [DOI] |
| 15. | Khor B, Gardet A, Xavier RJ. Genetics and pathogenesis of inflammatory bowel disease. Nature. 2011;474:307-317. [PubMed] [DOI] |
| 16. | Hou JK, Abraham B, El-Serag H. Dietary intake and risk of developing inflammatory bowel disease: a systematic review of the literature. Am J Gastroenterol. 2011;106:563-573. [PubMed] [DOI] |
| 17. | Jowett SL, Seal CJ, Pearce MS, Phillips E, Gregory W, Barton JR, Welfare MR. Influence of dietary factors on the clinical course of ulcerative colitis: a prospective cohort study. Gut. 2004;53:1479-1484. [PubMed] [DOI] |
| 18. | Ananthakrishnan AN, Khalili H, Konijeti GG, Higuchi LM, de Silva P, Korzenik JR, Fuchs CS, Willett WC, Richter JM, Chan AT. A prospective study of long-term intake of dietary fiber and risk of Crohn's disease and ulcerative colitis. Gastroenterology. 2013;145:970-977. [PubMed] [DOI] |
| 19. | Ananthakrishnan AN, Higuchi LM, Huang ES, Khalili H, Richter JM, Fuchs CS, Chan AT. Aspirin, nonsteroidal anti-inflammatory drug use, and risk for Crohn disease and ulcerative colitis: a cohort study. Ann Intern Med. 2012;156:350-359. [PubMed] [DOI] |
| 20. | Selby W, Pavli P, Crotty B, Florin T, Radford-Smith G, Gibson P, Mitchell B, Connell W, Read R, Merrett M, Ee H, Hetzel D; Antibiotics in Crohn's Disease Study Group. Two-year combination antibiotic therapy with clarithromycin, rifabutin, and clofazimine for Crohn's disease. Gastroenterology. 2007;132:2313-2319. [PubMed] [DOI] |
| 21. | Sartor RB, Wu GD. Roles for Intestinal Bacteria, Viruses, and Fungi in Pathogenesis of Inflammatory Bowel Diseases and Therapeutic Approaches. Gastroenterology. 2017;152:327-339.e4. [PubMed] [DOI] |
| 22. | Kvasnovsky CL, Aujla U, Bjarnason I. Nonsteroidal anti-inflammatory drugs and exacerbations of inflammatory bowel disease. Scand J Gastroenterol. 2015;50:255-263. [PubMed] [DOI] |
| 23. | Shorter RG, Huizenga KA, Spencer RJ. A working hypothesis for the etiology and pathogenesis of nonspecific inflammatory bowel disease. Am J Dig Dis. 1972;17:1024-1032. [PubMed] [DOI] |
| 24. | Chieppa M, Rescigno M, Huang AY, Germain RN. Dynamic imaging of dendritic cell extension into the small bowel lumen in response to epithelial cell TLR engagement. J Exp Med. 2006;203:2841-2852. [PubMed] [DOI] |
| 25. | Söderholm JD, Olaison G, Peterson KH, Franzén LE, Lindmark T, Wirén M, Tagesson C, Sjödahl R. Augmented increase in tight junction permeability by luminal stimuli in the non-inflamed ileum of Crohn's disease. Gut. 2002;50:307-313. [PubMed] [DOI] |
| 26. | Irvine EJ, Marshall JK. Increased intestinal permeability precedes the onset of Crohn's disease in a subject with familial risk. Gastroenterology. 2000;119:1740-1744. [PubMed] [DOI] |
| 27. | May GR, Sutherland LR, Meddings JB. Is small intestinal permeability really increased in relatives of patients with Crohn's disease? Gastroenterology. 1993;104:1627-1632. [PubMed] [DOI] |
| 28. | Muise AM, Walters TD, Glowacka WK, Griffiths AM, Ngan BY, Lan H, Xu W, Silverberg MS, Rotin D. Polymorphisms in E-cadherin (CDH1) result in a mis-localised cytoplasmic protein that is associated with Crohn's disease. Gut. 2009;58:1121-1127. [PubMed] [DOI] |
| 29. | Caruso R, Lo BC, Núñez G. Host-microbiota interactions in inflammatory bowel disease. Nat Rev Immunol. 2020;20:411-426. [PubMed] [DOI] |
| 30. | Ramos GP, Papadakis KA. Mechanisms of Disease: Inflammatory Bowel Diseases. Mayo Clin Proc. 2019;94:155-165. [PubMed] [DOI] |
| 31. | Corridoni D, Arseneau KO, Cominelli F. Inflammatory bowel disease. Immunol Lett. 2014;161:231-235. [PubMed] [DOI] |
| 32. | Kamada N, Hisamatsu T, Okamoto S, Chinen H, Kobayashi T, Sato T, Sakuraba A, Kitazume MT, Sugita A, Koganei K, Akagawa KS, Hibi T. Unique CD14 intestinal macrophages contribute to the pathogenesis of Crohn disease via IL-23/IFN-gamma axis. J Clin Invest. 2008;118:2269-2280. [PubMed] [DOI] |
| 33. | Fujino S, Andoh A, Bamba S, Ogawa A, Hata K, Araki Y, Bamba T, Fujiyama Y. Increased expression of interleukin 17 in inflammatory bowel disease. Gut. 2003;52:65-70. [PubMed] [DOI] |
| 34. | Teng MW, Bowman EP, McElwee JJ, Smyth MJ, Casanova JL, Cooper AM, Cua DJ. IL-12 and IL-23 cytokines: from discovery to targeted therapies for immune-mediated inflammatory diseases. Nat Med. 2015;21:719-729. [PubMed] [DOI] |
| 35. | Himmel ME, Yao Y, Orban PC, Steiner TS, Levings MK. Regulatory T-cell therapy for inflammatory bowel disease: more questions than answers. Immunology. 2012;136:115-122. [PubMed] [DOI] |
| 36. | O'Shea JJ, Schwartz DM, Villarino AV, Gadina M, McInnes IB, Laurence A. The JAK-STAT pathway: impact on human disease and therapeutic intervention. Annu Rev Med. 2015;66:311-328. [PubMed] [DOI] |
| 37. | Danese S, Panés J. Development of drugs to target interactions between leukocytes and endothelial cells and treatment algorithms for inflammatory bowel diseases. Gastroenterology. 2014;147:981-989. [PubMed] [DOI] |
| 38. | Arseneau KO, Cominelli F. Targeting leukocyte trafficking for the treatment of inflammatory bowel disease. Clin Pharmacol Ther. 2015;97:22-28. [PubMed] [DOI] |
| 39. | Vermeire S, O'Byrne S, Keir M, Williams M, Lu TT, Mansfield JC, Lamb CA, Feagan BG, Panes J, Salas A, Baumgart DC, Schreiber S, Dotan I, Sandborn WJ, Tew GW, Luca D, Tang MT, Diehl L, Eastham-Anderson J, De Hertogh G, Perrier C, Egen JG, Kirby JA, van Assche G, Rutgeerts P. Etrolizumab as induction therapy for ulcerative colitis: a randomised, controlled, phase 2 trial. Lancet. 2014;384:309-318. [PubMed] [DOI] |
| 40. | Jovani M, Danese S. Vedolizumab for the treatment of IBD: a selective therapeutic approach targeting pathogenic a4b7 cells. Curr Drug Targets. 2013;14:1433-1443. [PubMed] [DOI] |
| 41. | Eckburg PB, Relman DA. The role of microbes in Crohn's disease. Clin Infect Dis. 2007;44:256-262. [PubMed] [DOI] |
| 42. | Manichanh C, Borruel N, Casellas F, Guarner F. The gut microbiota in IBD. Nat Rev Gastroenterol Hepatol. 2012;9:599-608. [PubMed] [DOI] |
| 43. | Prosberg M, Bendtsen F, Vind I, Petersen AM, Gluud LL. The association between the gut microbiota and the inflammatory bowel disease activity: a systematic review and meta-analysis. Scand J Gastroenterol. 2016;51:1407-1415. [PubMed] [DOI] |
| 44. | Ohkusa T, Okayasu I, Ogihara T, Morita K, Ogawa M, Sato N. Induction of experimental ulcerative colitis by Fusobacterium varium isolated from colonic mucosa of patients with ulcerative colitis. Gut. 2003;52:79-83. [PubMed] [DOI] |
| 45. | Atarashi K, Tanoue T, Oshima K, Suda W, Nagano Y, Nishikawa H, Fukuda S, Saito T, Narushima S, Hase K, Kim S, Fritz JV, Wilmes P, Ueha S, Matsushima K, Ohno H, Olle B, Sakaguchi S, Taniguchi T, Morita H, Hattori M, Honda K. Treg induction by a rationally selected mixture of Clostridia strains from the human microbiota. Nature. 2013;500:232-236. [PubMed] [DOI] |
| 46. | Sartor RB. Microbial influences in inflammatory bowel diseases. Gastroenterology. 2008;134:577-594. [PubMed] [DOI] |
| 47. | Liao Z, Xie Y, Zhou B, Zou B, Xiao D, Liu W, Cai Y, Liu D, Liao Q, Xie Z. Berberine ameliorates colonic damage accompanied with the modulation of dysfunctional bacteria and functions in ulcerative colitis rats. Appl Microbiol Biotechnol. 2020;104:1737-1749. [PubMed] [DOI] |
| 48. | Liao Z, Zhang S, Liu W, Zou B, Lin L, Chen M, Liu D, Wang M, Li L, Cai Y, Liao Q, Xie Z. LC-MS-based metabolomics analysis of Berberine treatment in ulcerative colitis rats. J Chromatogr B Analyt Technol Biomed Life Sci. 2019;1133:121848. [PubMed] [DOI] |
| 50. | Hu J, Lin S, Zheng B, Cheung PCK. Short-chain fatty acids in control of energy metabolism. Crit Rev Food Sci Nutr. 2018;58:1243-1249. [PubMed] [DOI] |
| 52. | Bifari F, Ruocco C, Decimo I, Fumagalli G, Valerio A, Nisoli E. Amino acid supplements and metabolic health: a potential interplay between intestinal microbiota and systems control. Genes Nutr. 2017;12:27. [PubMed] [DOI] |
| 53. | Lin R, Liu W, Piao M, Zhu H. A review of the relationship between the gut microbiota and amino acid metabolism. Amino Acids. 2017;49:2083-2090. [PubMed] [DOI] |
| 54. | Lamas B, Richard ML, Leducq V, Pham HP, Michel ML, Da Costa G, Bridonneau C, Jegou S, Hoffmann TW, Natividad JM, Brot L, Taleb S, Couturier-Maillard A, Nion-Larmurier I, Merabtene F, Seksik P, Bourrier A, Cosnes J, Ryffel B, Beaugerie L, Launay JM, Langella P, Xavier RJ, Sokol H. CARD9 impacts colitis by altering gut microbiota metabolism of tryptophan into aryl hydrocarbon receptor ligands. Nat Med. 2016;22:598-605. [PubMed] [DOI] |
| 55. | Liu Y, Wang X, Hu CA. Therapeutic Potential of Amino Acids in Inflammatory Bowel Disease. Nutrients. 2017;9:920. [PubMed] [DOI] |
| 56. | Zheng X, Huang F, Zhao A, Lei S, Zhang Y, Xie G, Chen T, Qu C, Rajani C, Dong B, Li D, Jia W. Bile acid is a significant host factor shaping the gut microbiome of diet-induced obese mice. BMC Biol. 2017;15:120. [PubMed] [DOI] |
| 57. | Lamas B, Natividad JM, Sokol H. Aryl hydrocarbon receptor and intestinal immunity. Mucosal Immunol. 2018;11:1024-1038. [PubMed] [DOI] |
| 58. | Hooper LV, Gordon JI. Commensal host-bacterial relationships in the gut. Science. 2001;292:1115-1118. [PubMed] [DOI] |
| 59. | Wen Z, Fiocchi C. Inflammatory bowel disease: autoimmune or immune-mediated pathogenesis? Clin Dev Immunol. 2004;11:195-204. [PubMed] [DOI] |
| 60. | Gaboriau-Routhiau V, Rakotobe S, Lécuyer E, Mulder I, Lan A, Bridonneau C, Rochet V, Pisi A, De Paepe M, Brandi G, Eberl G, Snel J, Kelly D, Cerf-Bensussan N. The key role of segmented filamentous bacteria in the coordinated maturation of gut helper T cell responses. Immunity. 2009;31:677-689. [PubMed] [DOI] |
| 61. | Bauer E, Williams BA, Smidt H, Verstegen MW, Mosenthin R. Influence of the gastrointestinal microbiota on development of the immune system in young animals. Curr Issues Intest Microbiol. 2006;7:35-51. [PubMed] |
| 62. | Hapfelmeier S, Lawson MA, Slack E, Kirundi JK, Stoel M, Heikenwalder M, Cahenzli J, Velykoredko Y, Balmer ML, Endt K, Geuking MB, Curtiss R, McCoy KD, Macpherson AJ. Reversible microbial colonization of germ-free mice reveals the dynamics of IgA immune responses. Science. 2010;328:1705-1709. [PubMed] [DOI] |
| 63. | Zhu L, Xu LZ, Zhao S, Shen ZF, Shen H, Zhan LB. Protective effect of baicalin on the regulation of Treg/Th17 balance, gut microbiota and short-chain fatty acids in rats with ulcerative colitis. Appl Microbiol Biotechnol. 2020;104:5449-5460. [PubMed] [DOI] |
| 64. | Gutiérrez-Vázquez C, Quintana FJ. Regulation of the Immune Response by the Aryl Hydrocarbon Receptor. Immunity. 2018;19-33. [PubMed] [DOI] |
| 65. | Buffie CG, Pamer EG. Microbiota-mediated colonization resistance against intestinal pathogens. Nat Rev Immunol. 2013;13:790-801. [PubMed] [DOI] |
| 66. | Kamada N, Kim YG, Sham HP, Vallance BA, Puente JL, Martens EC, Núñez G. Regulated virulence controls the ability of a pathogen to compete with the gut microbiota. Science. 2012;336:1325-1329. [PubMed] [DOI] |
| 67. | Huang T, Zhang X, Pan J, Su X, Jin X, Guan X. Purification and Characterization of a Novel Cold Shock Protein-Like Bacteriocin Synthesized by Bacillus thuringiensis. Sci Rep. 2016;6:35560. [PubMed] [DOI] |
| 68. | Kinnebrew MA, Ubeda C, Zenewicz LA, Smith N, Flavell RA, Pamer EG. Bacterial flagellin stimulates Toll-like receptor 5-dependent defense against vancomycin-resistant Enterococcus infection. J Infect Dis. 2010;201:534-543. [PubMed] [DOI] |
| 69. | Brandl K, Plitas G, Mihu CN, Ubeda C, Jia T, Fleisher M, Schnabl B, DeMatteo RP, Pamer EG. Vancomycin-resistant enterococci exploit antibiotic-induced innate immune deficits. Nature. 2008;455:804-807. [PubMed] [DOI] |
| 70. | Jamwal S, Kumar P, Chapter 19 - animal models of inflammatory bowel disease, in Animal models for the study of human disease (second edition), P.M. Conn, Editor. 2017, Academic Press. 467-477. |
| 71. | Valatas V, Bamias G, Kolios G. Experimental colitis models: Insights into the pathogenesis of inflammatory bowel disease and translational issues. Eur J Pharmacol. 2015;759:253-264. [PubMed] [DOI] |
| 72. | Cooper HS, Murthy SN, Shah RS, Sedergran DJ. Clinicopathologic study of dextran sulfate sodium experimental murine colitis. Lab Invest. 1993;238-249. [PubMed] |
| 73. | Zheng H, Chen M, Li Y, Wang Y, Wei L, Liao Z, Wang M, Ma F, Liao Q, Xie Z. Modulation of Gut Microbiome Composition and Function in Experimental Colitis Treated with Sulfasalazine. Front Microbiol. 2017;8:1703. [PubMed] [DOI] |
| 74. | Cai Y, Liu W, Lin Y, Zhang S, Zou B, Xiao D, Lin L, Zhong Y, Zheng H, Liao Q, Xie Z. Compound polysaccharides ameliorate experimental colitis by modulating gut microbiota composition and function. J Gastroenterol Hepatol. 2019;34:1554-1562. [PubMed] [DOI] |
| 75. | Kotlarz D, Beier R, Murugan D, Diestelhorst J, Jensen O, Boztug K, Pfeifer D, Kreipe H, Pfister ED, Baumann U, Puchalka J, Bohne J, Egritas O, Dalgic B, Kolho KL, Sauerbrey A, Buderus S, Güngör T, Enninger A, Koda YK, Guariso G, Weiss B, Corbacioglu S, Socha P, Uslu N, Metin A, Wahbeh GT, Husain K, Ramadan D, Al-Herz W, Grimbacher B, Sauer M, Sykora KW, Koletzko S, Klein C. Loss of interleukin-10 signaling and infantile inflammatory bowel disease: implications for diagnosis and therapy. Gastroenterology. 2012;143:347-355. [PubMed] [DOI] |
| 76. | Britton GJ, Contijoch EJ, Mogno I, Vennaro OH, Llewellyn SR, Ng R, Li Z, Mortha A, Merad M, Das A, Gevers D, McGovern DPB, Singh N, Braun J, Jacobs JP, Clemente JC, Grinspan A, Sands BE, Colombel JF, Dubinsky MC, Faith JJ. Microbiotas from Humans with Inflammatory Bowel Disease Alter the Balance of Gut Th17 and RORγt+ Regulatory T Cells and Exacerbate Colitis in Mice. Immunity. 2019;50:212-224.e4. [PubMed] [DOI] |
| 77. | Gobert AP, Sagrestani G, Delmas E, Wilson KT, Verriere TG, Dapoigny M, Del'homme C, Bernalier-Donadille A. The human intestinal microbiota of constipated-predominant irritable bowel syndrome patients exhibits anti-inflammatory properties. Sci Rep. 2016;6:39399. [PubMed] [DOI] |
| 78. | Cui H, Cai Y, Wang L, Jia B, Li J, Zhao S, Chu X, Lin J, Zhang X, Bian Y, Zhuang P. Berberine Regulates Treg/Th17 Balance to Treat Ulcerative Colitis Through Modulating the Gut Microbiota in the Colon. Front Pharmacol. 2018;9:571. [PubMed] [DOI] |
| 79. | Elinav E, Strowig T, Kau AL, Henao-Mejia J, Thaiss CA, Booth CJ, Peaper DR, Bertin J, Eisenbarth SC, Gordon JI, Flavell RA. NLRP6 inflammasome regulates colonic microbial ecology and risk for colitis. Cell. 2011;145:745-757. [PubMed] [DOI] |
| 80. | Rakoff-Nahoum S, Paglino J, Eslami-Varzaneh F, Edberg S, Medzhitov R. Recognition of commensal microflora by toll-like receptors is required for intestinal homeostasis. Cell. 2004;118:229-241. [PubMed] [DOI] |
| 81. | Zhao L, Yang W, Chen Y, Huang F, Lu L, Lin C, Huang T, Ning Z, Zhai L, Zhong LL, Lam W, Yang Z, Zhang X, Cheng C, Han L, Qiu Q, Shang X, Huang R, Xiao H, Ren Z, Chen D, Sun S, El-Nezami H, Cai Z, Lu A, Fang X, Jia W, Bian Z. A Clostridia-rich microbiota enhances bile acid excretion in diarrhea-predominant irritable bowel syndrome. J Clin Invest. 2020;130:438-450. [PubMed] [DOI] |
| 82. | Davido B, Batista R, Dinh A, de Truchis P, Terveer EM, Roberts B, Kuijper EJ, Caballero S. Fifty shades of graft: How to improve the efficacy of faecal microbiota transplantation for decolonization of antibiotic-resistant bacteria. Int J Antimicrob Agents. 2019;53:553-556. [PubMed] [DOI] |
| 83. | Giles EM, D'Adamo GL, Forster SC. The future of faecal transplants. Nat Rev Microbiol. 2019;17:719. [PubMed] [DOI] |
| 84. | Bamola VD, Ghosh A, Kapardar RK, Lal B, Cheema S, Sarma P, Chaudhry R. Gut microbial diversity in health and disease: experience of healthy Indian subjects, and colon carcinoma and inflammatory bowel disease patients. Microb Ecol Health Dis. 2017;28:1322447. [PubMed] [DOI] |
| 85. | Dickson I. Gut microbiota: Diagnosing IBD with the gut microbiome. Nat Rev Gastroenterol Hepatol. 2017;14:195. [PubMed] [DOI] |
| 86. | Sheil B, Shanahan F, O'Mahony L. Probiotic effects on inflammatory bowel disease. J Nutr. 2007;137:819S-824S. [PubMed] [DOI] |
| 87. | Sartor RB. Therapeutic manipulation of the enteric microflora in inflammatory bowel diseases: antibiotics, probiotics, and prebiotics. Gastroenterology. 2004;126:1620-1633. [PubMed] [DOI] |
| 88. | Chen H, Xia Y, Zhu S, Yang J, Yao J, Di J, Liang Y, Gao R, Wu W, Yang Y, Shi C, Hu D, Qin H, Wang Z. Lactobacillus plantarum LP-Onlly alters the gut flora and attenuates colitis by inducing microbiome alteration in interleukin-10 knockout mice. Mol Med Rep. 2017;16:5979-5985. [PubMed] [DOI] |
| 89. | Schultz M, Veltkamp C, Dieleman LA, Grenther WB, Wyrick PB, Tonkonogy SL, Sartor RB. Lactobacillus plantarum 299V in the treatment and prevention of spontaneous colitis in interleukin-10-deficient mice. Inflamm Bowel Dis. 2002;8:71-80. [PubMed] [DOI] |
| 90. | Lopez-Siles M, Enrich-Capó N, Aldeguer X, Sabat-Mir M, Duncan SH, Garcia-Gil LJ, Martinez-Medina M. Alterations in the Abundance and Co-occurrence of Akkermansia muciniphila and Faecalibacterium prausnitzii in the Colonic Mucosa of Inflammatory Bowel Disease Subjects. Front Cell Infect Microbiol. 2018;8:281. [PubMed] [DOI] |